Для этого пратикума я использовала бактерии из праткикума 1. Я загрузила протеомы этих бактерий в один файл, а затем проиндексировала последовательности командой makeblastdb -dbtype prot -in proteomes_pr1.fasta. С помощью команды blastp -query clpx_ecoli.fasta -db proteomes.fasta -evalue 0.001 -out proteomes_blastp, были найдены достоверные гомологи CLPX_ECOLI.
С сайта Uniprot я скачала последовательности нужных белков. Затем программой mucsle я выровняла их. Методом N-J в программе MEGAX я построила филогенетическое дерево.
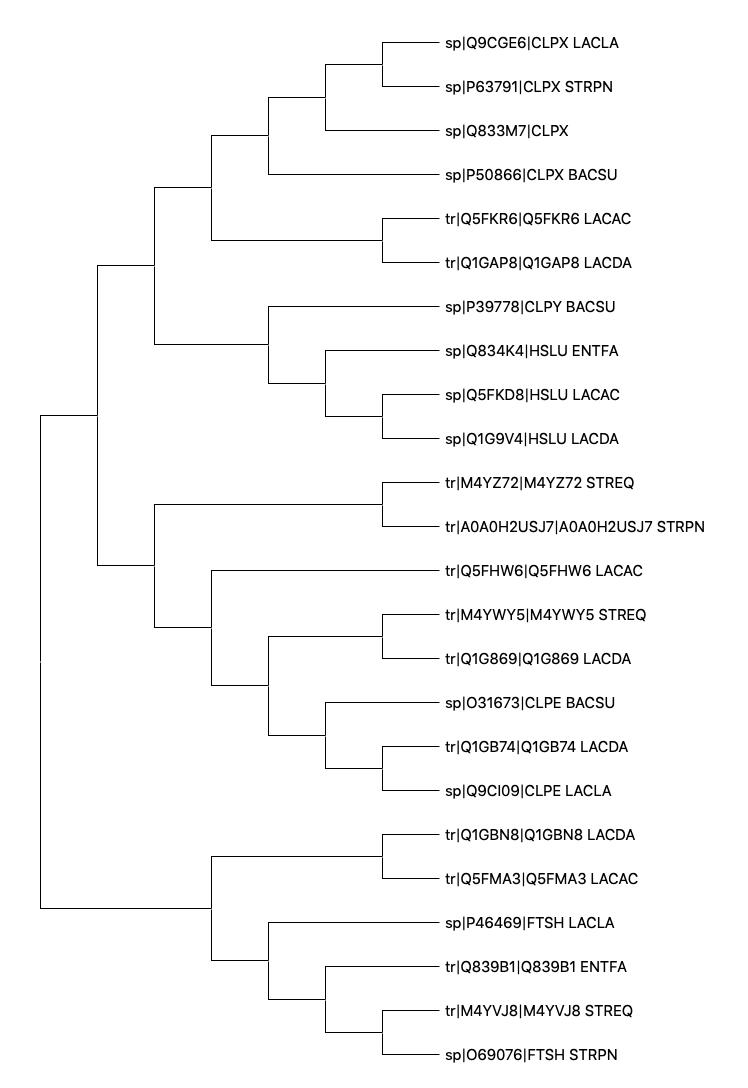
Получившееся дерево.

Примеры пар ортологов: CLPX_LACLA-CLPX_ENTFA, HSLU_LACAC-ENTFA, FTSH_STRPN-FTSH_LACLA
Примеры пар паралогов: Q1G869 LACDA и Q1GB74 LACDA, CLPX ENTFA и HSLU ENTFA, Q5FKR6 LACAC и HSLU LACAC.
Дерево с выделенными группами ортологов.

Дерево со схлопнутыми группами. Для всех групп филогения соостветствует филогении бактерий.